代谢组学的原始数据
刚拿到代谢组的数据
刚拿到代谢组的数据一头雾水,打开excel一看是密密麻麻的数据,不知从何入手。
这个笔记是用来记录分析代谢组数据的步骤。
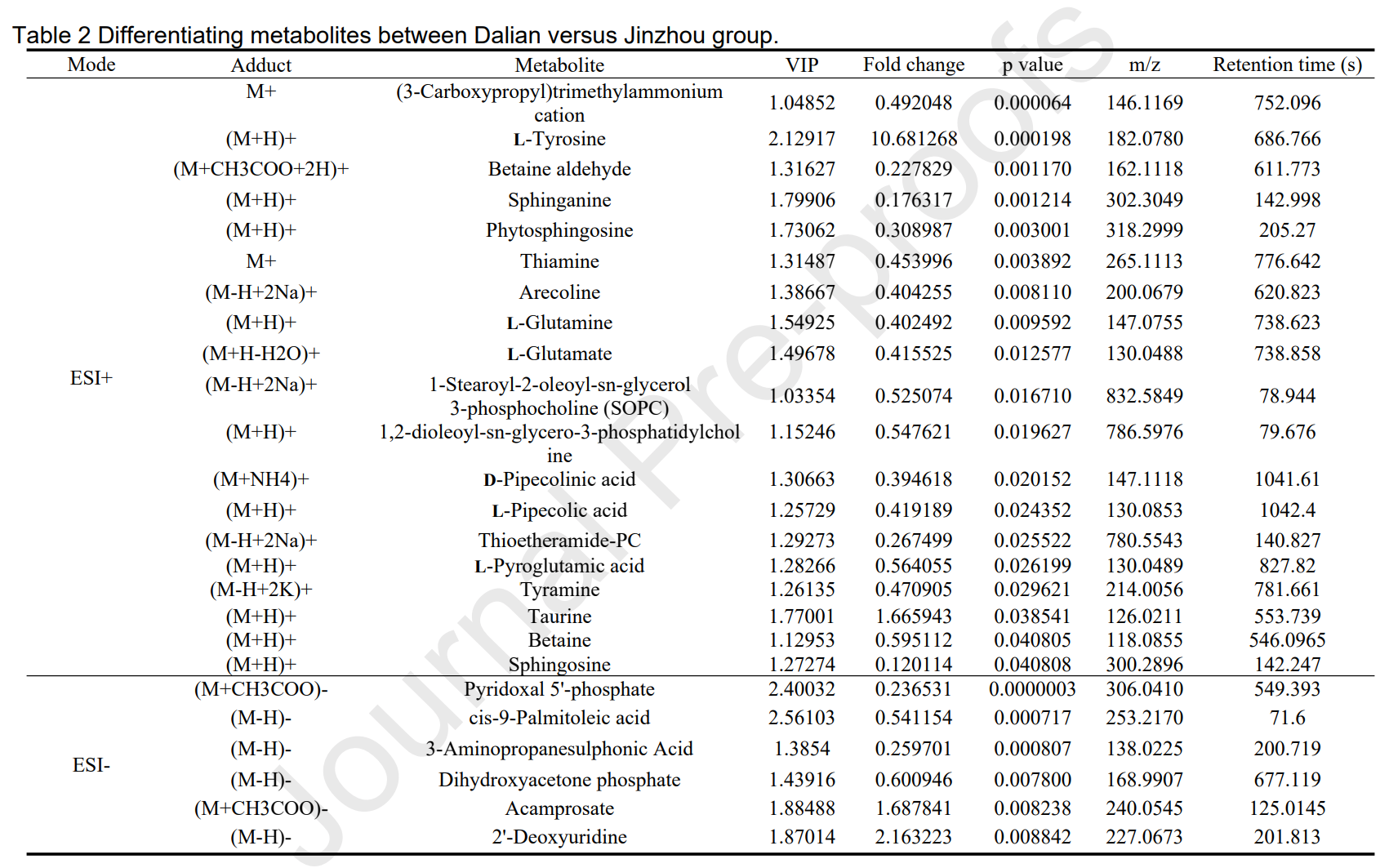
直观数据上,首先最重要的参数有:信号强度,P值(如果没有分析好,需要用t检验分析)。
把P>0.05的数据行都删掉,把没有识别出代谢物的行也删掉。
找到你的分组,举例来说,一般有空白对照组、模型组、实验组。
将每组取均值,用均值计算组间的差异倍数(FC)。
一般而言,组间比较有:模型组比空白对照组、实验组比模型组。
那么,模型组比空白对照组的差异倍数,FC=模型均值/空白组均值。
再取其以2为底的log对数,获得log2FC。有的数据分析以FC判别,有的以log2FC判别,这并无差异。
根据P值计算FDR,也是Padj,后面的筛选一般用这个代替P值。
FDR=false discovery rate(伪发现率),计算方法:
- 对P值排序,从小到大
- FDR=P×总数÷排序号
现在我们有了FC、log2FC、FDR,就可以进行下一步了。
差异代谢物可视化
最后要呈现的数据
ORA富集分析和SMPDB通路分析
使用MetaboAnalyst 6.0网页版基于SMPDB数据库进行ORA富集分析(Enrichment Analysis)和通路分析(Pathway Analysis)。
将VIP大于1的代谢化合物HMDB编号输入网站数据框,执行过度表示分析(Over Representation Analysis, ORA)。ORA是使用超几何测试来评估特定代谢物组在给定化合物列表中的表现是否超出预期。在对多次测试进行调整后,提供单尾P值。将代谢物映射到生物通路,了解代谢通路的变化情况。
